
Kontrola na úrovni sond
Tato kontrola se neprovádí standardně pro všechny sondy, ale nejčastěji v případě, že potřebujeme vědět, zdali je určitá sada sond funkční ve smyslu správné reprezentace cílové sekvence. Můžeme si zobrazit PM intensity sond v sadě s identifikačním číslem "240060_at":
> pm(Data,"240060_at")
1 2 3 4 5 6 7 14
240058_at1 78.0 59.3 50.5 74.0 70.0 53.8 85.3 136.8
240058_at2 69.5 85.8 47.0 84.5 58.5 53.3 78.3 178.0
240058_at3 103.5 116.3 57.0 101.5 80.5 65.3 137.5 201.0
240058_at4 65.8 146.0 52.3 71.3 63.3 62.3 88.5 155.3
240058_at5 102.0 153.3 58.0 81.3 86.3 65.3 128.3 193.0
240058_at6 92.0 80.3 67.0 89.0 74.0 67.8 123.0 211.3
240058_at7 120.3 83.5 73.8 94.8 89.5 88.3 168.3 246.8
240058_at8 82.0 53.8 44.0 59.8 62.3 59.5 86.0 146.3
240058_at9 79.3 123.3 49.3 80.0 76.0 59.0 107.3 210.5
240058_at10 290.0 237.8 122.0 198.8 185.5 138.0 323.3 598.0
240058_at11 166.3 104.3 83.5 123.3 132.0 107.3 291.8 373.0
Funkce matplot vykreslí automaticky intensity sond podle jejich umístnění od 5’ ke 3’ konci sekvence transkriptu, který představují (obrázek 4.1). Například sada sond 240060_at z MLL datového souboru obsahuje pouze jednu sondu, která vykazuje dostatečně vysokou intensitu hybridizace. To může znamenat, že ostatní sondy sady nebyly dobře navrženy, nebo že právě u této sondy došlo k nespecifické hybridizaci, což se dá zjistit inspekcí MM hodnot sondy:
> par(mfrow=c(1,2))
> matplot(pm(Data,"240060_at"), type="l", ylab="PM intensita sondy", xlab="Sada sond 240060_at", las=1, main="PM")
> matplot(mm(Data,"240060_at"), type="l", ylab="MM intensita sondy", xlab="Sada sond 240060_at", las=1, main="MM"))
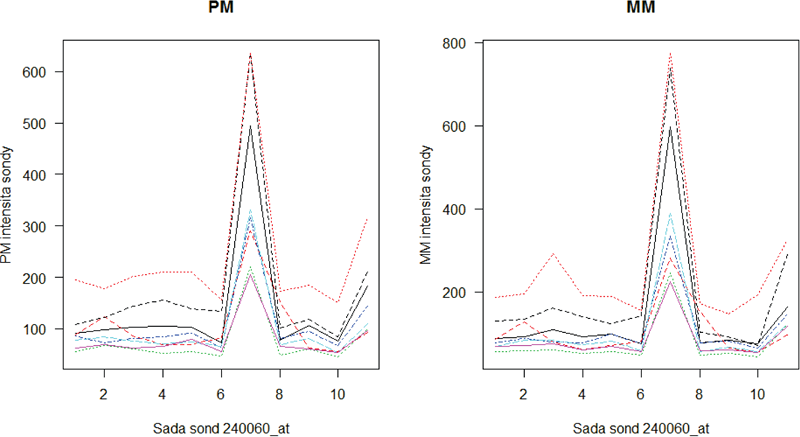
Obr. 4.1: Zobrazení PM (vlevo) a MM (vpravo) intensit sond ze sady 240060_at. Jednotlivé křivky představují mikročipy. Pouze sonda č. 7 má vysokou expresi u všech mikročipů, nicméně zřejmě je to důsledek nespecifické hybridizace.
Dalším využitím může být vizuální porovnání exprese jednotlivých sond mezi dvěma skupinami. Ukázkovým příkladem bude sada sond "205225_at" reprezentující gen pro ESR1 protein, který rozlišuje ER + a ER- skupinu nádoru prsu (obrázek 4.2). Použijeme soubor breast:
> par(mfrow=c(1,2))
> matplot(pm(Xraw,"205225_at"), type="l", ylab="PM intensita sondy", xlab="Sada sond 205225_at", las=1, col=as.numeric(sampleInfo$ER_status), main="ESR1, PM", ylim=c(0,13000))
> matplot(mm(Xraw,"205225_at"), type="l", ylab="MM intensita sondy", xlab="Sada sond 205225_at", las=1, col=as.numeric(sampleInfo$ER_status), main="ESR1, MM", ylim=c(0,13000))
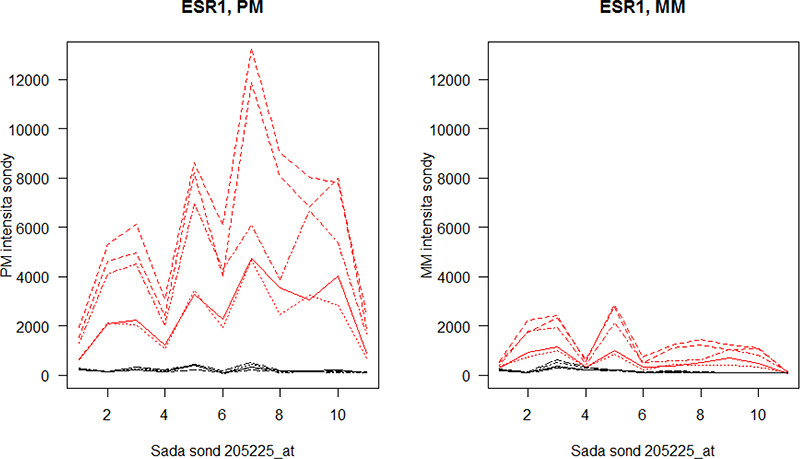
Obr. 4.2: Zobrazení PM (vlevo) a MM (vpravo) intensit sond ze sady sond 205225_at, reprezentující gen pro ESR1 protein. Jednotlivé křivky představují mikročipy, černé křivky představují ER+ a červené pak ER- nádory prsu.




