
Praktický příklad analýzy
Ukažme si na příkladu jak může probíhat analýza přežití v analýze genomických a proteomických dat.
Budeme pokračovat v práci se stejným příkladem experimentu porovnávajícího ER (estrogen receptor) pozitivní a ER negativní karcinomy prsu, který jsme používali v předcházejících kapitolách. Budeme používat výsledky z praktického příkladu analýzy z kapitoly "Porovnávání skupin" a to zejména:
- objekt top100 obsahující top 100 odlišně exprimovaných genů mezi skupinami ER+ a ER- (jak získat top100 najdete ZDE)
Načteme si nejdříve genové expresní data karcinomu prsu TransBig spolu s tabulkou klinických dat. Data lze stáhnout ZDE (nutno rozbalit archiv). Načteme také top 100 genů odlišně
> load(file="TransBIG/TransBIG-expression.rdata")
> clinical2 <- read.csv("TransBIG/TransBIG-Sample_info.csv")
> load("top100.rdata")
1) Chceme zjistit rozdíl v přežití mezi skupinami ER+ a ER-
Načteme funkci pro vykreslení Kaplan-Meierových křivek a balík pro analýzu přežití:
> source("surv.plot.R")
> library(survival)
Loading required package: splines
> rfs <- clinical2$Time.RFS..days
> rfs.event <- clinical2$Event.RFS
> surv.plot(survfit(Surv(rfs, rfs.event)~clinical2$ER.pos.1.yes.))
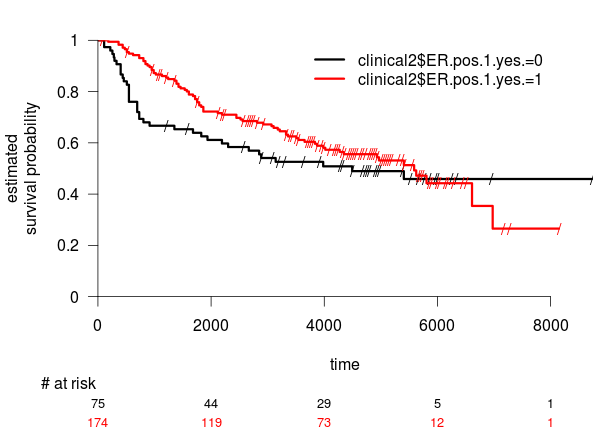
Odhadneme medián přežití:
> survfit(Surv(rfs, rfs.event)~clinical2$ER.pos.1.yes.)
Call: survfit(formula = Surv(rfs, rfs.event) ~ clinical2$ER.pos.1.yes.)
4 observations deleted due to missingness
records n.max n.start events median 0.95LCL 0.95UCL
clinical2$ER.pos.1.yes.=0 75 75 75 38 4493 2192 NA
clinical2$ER.pos.1.yes.=1 174 174 174 79 5588 4273 NA
Je rozdíl v přežití statisticky významný?
> survdiff(Surv(rfs, rfs.event)~clinical2$ER.pos.1.yes.)
Call:
survdiff(formula = Surv(rfs, rfs.event) ~ clinical2$ER.pos.1.yes.)
n=249, 4 observations deleted due to missingness.
N Observed Expected (O-E)^2/E (O-E)^2/V
clinical2$ER.pos.1.yes.=0 75 38 31.8 1.208 1.67
clinical2$ER.pos.1.yes.=1 174 79 85.2 0.451 1.67
Chisq= 1.7 on 1 degrees of freedom, p= 0.196
Není, i když je tam trend. Nyní to zkusíme se skupinami definovanými shlukováním, kterým jsme v kapitole Objevování skupin tvořili na datech heatmapu. Heatmapa sice vnitřně aplikovala shlukovací algoritmus, ale my to musíme udělat znovu abychom získali dendrogram, ze kterého pak vyextrahujeme příslušnost ke skupinám.
> eukl2 <- dist(X[,intersect(colnames(X),top100)])
> hclust2 <- hclust(eukl2)
> plot(hclust2)

> groups2 <- cutree(hclust2, k=2)# delime na dve skupiny
> head(groups2)
4000471 4000473 4000475 4000477 4000480 4000482
1 2 2 2 2 1
Nyní analýza přežití s novými skupinami:
> surv.plot(survfit(Surv(rfs, rfs.event)~groups2))
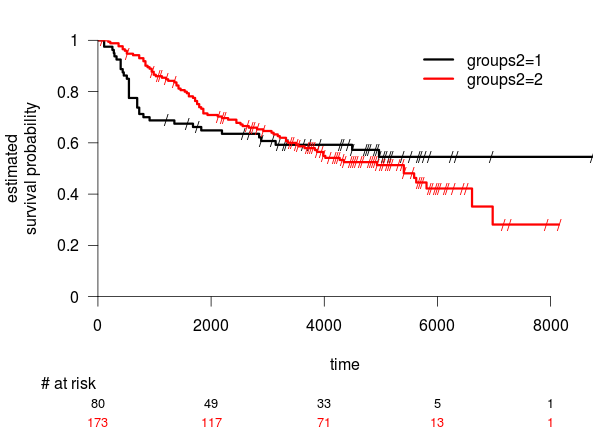
Další úlohy k procvičení:
1) Udělejte podobnou analýzu s top200 a top500 geny
2) Poté udělejte podobnou analýzu s HER2
Nyní vše zkusíme spojit. Modelování pomocí Coxova modelu. Aplikujeme normalizaci, median clustering a IQR dělení.
Vypočítáme madián a IQR exprese každého genu.
> med<-median(X)
> iqr<-IQR(X)
> X2<-(X-med)/iqr
Má gen ESR1 nějaký vliv na dobu přežití do relapsu (RFS)?
> coxph(Surv(rfs, rfs.event)~X2[,"ESR1"])
Call:
coxph(formula = Surv(rfs, rfs.event) ~ X2[, "ESR1"])
coef exp(coef) se(coef) z p
X2[, "ESR1"] -0.00414 0.996 0.0231 -0.179 0.86
Likelihood ratio test=0.03 on 1 df, p=0.858 n= 253, number of events= 118
ESR1 nemá vliv na RFS (p = 0.86)
Nyní hledáme geny, které mají významný vliv na relepse-free-survival:
> surv.res<-c()
> for (i in c(1:ncol(X2)))
+ {
+ temp<-coxph(Surv(rfs, rfs.event)~X2[,i])
+ a<-summary(temp)
+ surv.res<-rbind(surv.res, c(colnames(X2)[i],a$coefficients))
+ }
> colnames(surv.res)<-c("gene",colnames(a$coefficients))




