
Kontrola kvality na základě modelu úrovně sondy (PLM - probe level model)
Tento typ kontroly kvality staví na lineárním modelu - intensit normalizovaných na pozadí pomocí RMA (viz níže), který se nazývá PLM model a je definován následovně:
|
|
(4.1)
|
kde představuje logaritmovanou hladinu exprese transkriptu (genu) g na mikročipu i,
je efekt k-té sondy reprezentující transkript g a
je chyba měření.
je tedy již sumarizovaná hodnota signálu všech sond ze sady reprezentující transcript g a odhaduje se buď pomocí mediánového vyhlazování (viz níže, u metody RMA), nebo pomocí robustní lineární regrese, jak je implementováno v balíku affyPLM. Tento balík poskytuje sadu funkcí pro kontrolu kvality mikročipů, využívajících odhad
, směrodatnou chybu tohoto odhadu
a rezidua.
> library(affyPLM)
Funkce, která odhaduje PLM model, vypočítá RMA normalizaci na pozadí automaticky, takže vstupní objekt je nenormalizovaný soubor základních matic intensit AffyBatch. Výsledkem je objekt třídy PLMset, který je stejný jako AffyBatch, pouze obsahuje další komponenty, jako jsou váhy, rezidua, odhady standardní chyby a další informace související s robustním modelem. Tyto komponenty pak slouží k lepší vizualizaci a charakterizaci dat, než samotné intensity signálů.
Pro ilustraci si vykreslíme a uložíme heatmapy původních intensit, váh, reziduí a znamének reziduí u našeho příkladového souboru MLL.
> PLMres = fitPLM(Data)
> par(mfrow=c(2,2), mar=c(1,1,3,1))
> for (i in c(1:8))
+ {
+ image(Data[,i], main="intensita signálu")
+ image(PLMres, type="weights", which=i, main="váhy")
+ image(PLMres, type="resids", which=i, main="rezidua")
+ image(PLMres, type="sign.resids", which=i, main="znaménka reziduí")
+ savePlot(file=paste("MLL_PLM_",i,".png",sep=""), type="png")
+ }
Inspekce výsledných grafů nám odhalí jednak očekávaný prostorový efekt u čipu 2, ale navíc i prostorové efekty u čipu 1, 4 nebo 7 (obrázek 4.5), které by nebyly patrny z heatmapy intensit.

Obr 4.6: Různé zobrazení virtuální rekonstrukce mikročipového sklíčka pro odhalení prostorových efektů s pomocí PLM modelu u MLL datového souboru, čip 7.
RLE a NUSE
Další dvě metriky vypočítané na základě odhadnutých parametrů z PLM modelu slouží jako rozhodovací nástroj o nekvalitě mikročipového sklíčka. Jsou to metriky RLE (relativní logaritmovaná exprese) a NUSE (normalizovaná neškálovaná standardní chyba), definované následovně:
|
|
(4.2) |
|
|
(4.3) |
Kde je odhad logaritmované hladiny exprese transkriptu (genu) g na mikročipu i, a
je medián těchto odhadů přes všechny mikročipy.
RLE tedy měří odchylku odhadu exprese transkriptu od mediánu těchto odhadů na všech čipech. Předpokládáme-li, že většina transkriptů na čipu nemá změněnou expresi, RLE hodnoty by se neměly výrazně lišit od nuly. NUSE je mediánem normalizovaná standardní chyba odhadu exprese transkriptu, a proto by se měla pohybovat kolem 1.
Pokud vzhledem k druhu experimentu a mikročipu můžeme očekávat, že platí předpoklad o nezměněné expresi většiny transkriptů, můžeme odstranit čip jako nekvalitní, pokud má výrazně posunuté RLE hodnoty mimo 0, a NUSE hodnoty nad 1. Na základě našich vlastních zkušeností doporučujeme vyřazovat mikročipy s mediánem NUSE >1.02:
> nuse.stat = nuse(PLMres, type="stats")
> W = nuse.stat["median",]<1.02
> W
1 2 3 4 5 6 7 14
TRUE FALSE TRUE TRUE TRUE TRUE TRUE TRUE
> Data.clean = Data[,W]
Funkce Mbox vykreslí krabicové grafy RLE hodnoty pro všechny čipy a funkce NUSE vykreslí krabicové grafy hodnot NUSE (obrázek 4.7):
> par(mfrow=c(2,2))
> Mbox(PLMres, main="RLE", las=1)
> abline(h=0, lty="dashed")
> s = nuse(PLMres, type="stats")
> NUSE(PLMres, ylim=c(0.9,2), las=1, main="NUSE")
> title("NUSE")
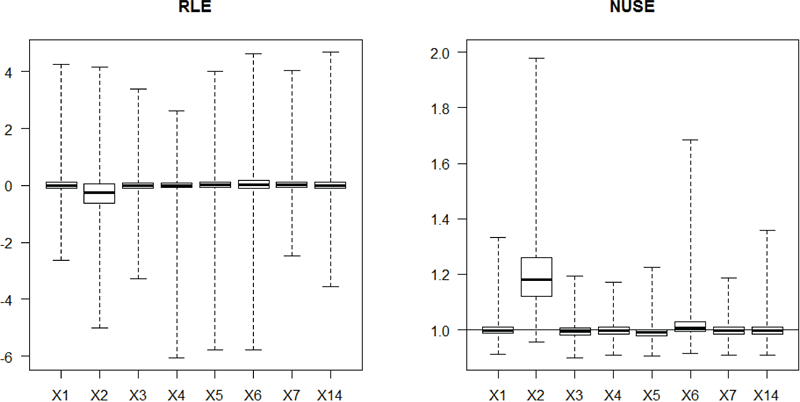
Obr. 4.7: Krabicové grafy RLE a NUSE metrik.
Obě hodnoty jsou výrazně posunuté u čipu 2, který vykazuje výrazným prostorový efekt.




