
Část 3: Skupiny krajů a plodin na základě výnosů
Kolik skupin krajů a plodin můžeme rozlišit na základě výnosů?
K tomuto účelu použijeme shlukovací analýzu. Konkrétně hierarchické shlukování, s použitím funkce hclust() z balíku cluster.
Základní použití funkce je následující:
hclust(d, method = "complete"), kde d= je matice vzdáleností mezi objekty, method= je metoda: "ward", "single", "complete", "average", "mcquitty", "median" alebo "centroid".
Výstupem je objekt třídy hclust, pro který existují zvláštní výstupy generických funkcí print(), plot(), atd.
Nyní chceme vědět, které ze zkoumaných plodin tvoří společně shluky na základě nutričního složení. Také chceme vědět, jestli zkoumané plodiny dokážou rozdělit kraje. Uděláme proto hierarchické shlukování jak plodin, tak krajů. Plodiny můžeme shlukovat buď na základě jejich výnosů, nebo na základě jejich nutričních skupin. Z předešlých dvou kroků analýzy víme, že můžeme očekávat tři skupiny plodin na základě nutričních skupin a také, že nejsou rozdíly ve výnosech mezi kraji. To naznačuje, že asi nenajdeme stabilní rozdělení krajů do shluků podle výnosů plodin.
Nejdříve pojmenujeme plodiny i kraje podle jejich názvů v tabulce uroda:
> colnames(uroda)<-plodiny[,1]
> rownames(uroda)<-kraje$Name
Pokud chceme shlukovat, nejprve musíme definovat matici vzdáleností d, která je základním vstupem do funkce hclust(). Použijeme dva druhy vzdálenosti:
1. Euklidovskou vzdálenost - funkce dist()
dist(x,method)
argumenty: x – datová tabulka, objekty mezi kterými počítáme vzdálenost musí být v řádcích
method – metrika vzdálenosti
> euclid.plodina <- dist(t(uroda), method="euclidean") # vzdálenost mezi plodinami
> euclid.kraj <- dist(uroda, method="euclidean") # vzdálenost mezi kraji
2. Vzdálenost založenou na Spearmanově korelaci - funkce cor()
cor(x,method) vypočítá korelaci mezi sloupci matice
argumenty: x – matice
method – "pearson" nebo "spearman"
> cor.nut <- cor(plodiny[,c(6:9)], method="spearman")#počítáme korelaci mezi výživovými hodnotami u plodin
> cor.nut
Karbohydráty Vláknina Tuky Bílkoviny
Karbohydráty 1.0000000 0.8618790 0.3156733 0.6909492
Vláknina 0.8618790 1.0000000 0.3668511 0.7292822
Tuky 0.3156733 0.3668511 1.0000000 0.5805740
Bílkoviny 0.6909492 0.7292822 0.5805740 1.0000000
> cor.plodina <- cor(uroda, method="spearman")#počítáme korelaci mezi plodinami na základě výnosů
> cor.kraj <- cor(t(uroda), method="spearman") #počítáme korelaci mezi kraji, na základě výnosů plodin
Potřebujeme ale vzdálenost, proto korelaci odečteme od 1 (korelace 1 bude vzdálenost 0 a korelace -1 bude vzdálenost 2) a použijeme funkci as.dist(), která nám matici korelací přemění na objekt typu dist, který potřebujeme do hclust() funkce.
> cor.plodina.dist <- as.dist(1-cor.plodina)
> cor.kraj.dist <- as.dist(1-cor.kraj)
> cor.nut.dist <- as.dist(1-cor.nut)
> cor.nut.dist
Karbohydráty Vláknina Tuky
Vláknina 0.1381210
Tuky 0.6843267 0.6331489
Bílkoviny 0.3090508 0.2707178 0.4194260
Teď již můžeme shlukovat plodiny na základě výnosů krajů - vybereme algoritmus average linking:
...na základě euklidovské vzdálenosti:
> hclust.plodina.eucl.avg <- hclust(euclid.plodina, method="average")
...a na vzdálenosti odvozené od korelace:
> hclust.plodina.spear.avg <- hclust(cor.plodina.dist, method="average")
Podobně shlukujeme kraje na základě výnosů plodin:
...na základě euklidovské vzdálenosti:
> hclust.kraj.eucl.avg <- hclust(euclid.kraj, method="average")
...a na vzdálenosti odvozené od korelace:
> hclust.kraj.spear.avg <- hclust(cor.kraj.dist, method="average")
Pro plodiny vykreslíme výsledky shlukování ve formě tzv dendrogramu pro obě vzdálenosti:
> png("dendrogram.plodina.png", width=480, height=600)
> par(mfrow=c(2,1))
> plot(hclust.plodina.eucl.avg)
> plot(hclust.plodina.spear.avg)
> dev.off()
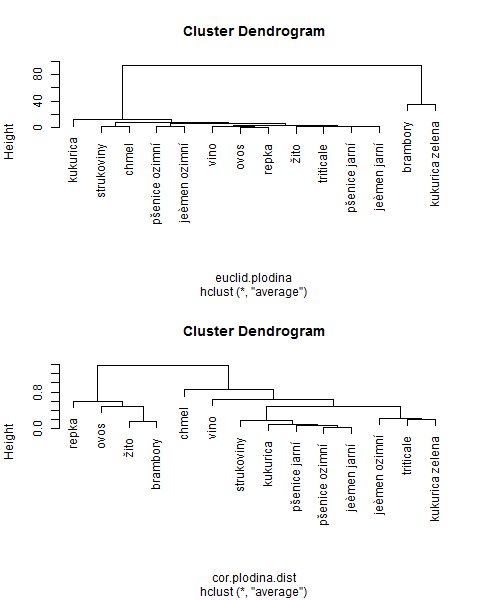
Vypadá to, že máme dva shluky, každá vzdálenost dává shluky jiné, což je v pořádku, protože každá ze vzdáleností měří něco jiného. Které plodiny tyto shluky obsahují sice můžeme odečíst, ale tuto informaci můžeme potřebovat pro další část analýzy ve vektorovém formátu. Použijeme proto funkci cutree() na objekt hclust a získáme zařazení plodin do skupin:
> grps_plant_eucl <- cutree(hclust.plant.eucl.avg, k=2)
> grps_plant_cor <- cutree(hclust.plant.spear.avg, k=2)
Zařazení do skupin můžeme přidat jako novou proměnnou do tabulky plodiny:
> plodiny$grps_plant <- grps_plant_eucl
> plodiny$grps_plant_cor <- grps_plant_cor
Totéž zopakujeme pro kraje:
> png("dendrogram.kraj.png", width=480, height=600)
> par(mfrow=c(2,1), cex=0.8)
> plot(hclust.kraj.eucl.avg)
> plot(hclust.kraj.spear.avg)
> dev.off()
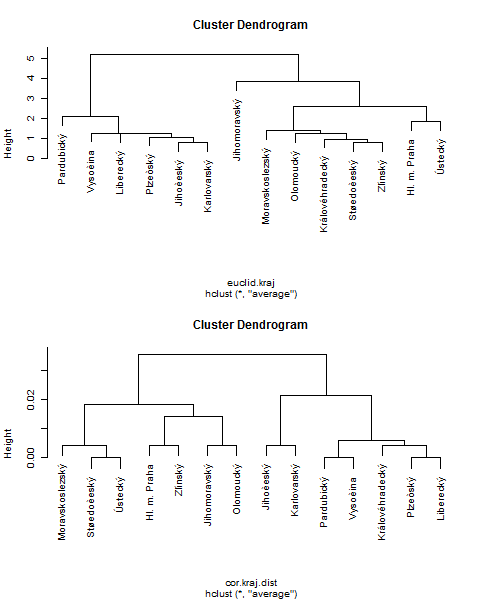
Znovu vidíme dvě skupiny a rozdíly v obou metrikách. Aplikujeme funkci cutree() pro získání přislušnosti ke skupině.
> grps_kraj_eucl <- cutree(hclust.kraj.eucl.avg, k=2)
> grps_kraj_cor <- cutree(hclust.kraj.spear.avg, k=2)
Teď můžeme porovnat obě shlukování:
> table(grps_kraj_eucl, grps_kraj_cor)
grps_kraj_cor
grps_kraj_eucl 1 2
1 7 1
2 0 6
Rozdíly mezi skupinami získanými shlukováním podle obou metrik jsou minimální. Opět přidáme nové proměnné do tabulky kraje:
> kraje$grps_kraj <- grps_kraj_eucl
> kraje$grps_kraj_cor <- grps_kraj_cor
Můžeme se teď podívat, jestli nové skupiny korelují například s regionem:
> table(kraje$grps_kraj_cor, kraje$Region)
Cechy Morava
1 3 4
2 7 0
Zdá se, že jedna ze dvou skupin obsahuje pouze české kraje.
Dalším krokem je vykreslení tzv. heatmapy, jak si ukážeme v další kapitole.




