
Část 1: Výnos v Čechách a na Moravě
Mají jednotlivé druhy plodin stejný výnos v Čechách a na Moravě? Pokud ne, u kterých plodin se výnosy liší?
Provedeme testování hypotéz
Informace o výnosu plodin je uložena v souboru uroda. Plodiny jsou uložené jako sloupce (i-tá plodina): uroda[,i]. Informace o příslušnosti krajů k regionu (Čechy versus Morava) je v souboru kraje. Uložíme si informaci o skupině do proměnné group.
> group <- kraje$Region
Který statistický test použijeme záleží na tom, jestli mají hektarové výnosy normální rozložení (u každého z regionů) nebo ne. V případě, že ano, použijeme parametrický T-test, v případě, že ne, použijeme na všechny plodiny Wilcoxnův neparametrický test.
Pro otestování normality použijeme Shapiro-Wilkův test shapiro.test(). Použijeme funkci apply() a tapply() pro testování každé z plodin v každé skupině:
> stres<-apply(as.matrix(uroda), 2, function(x) tapply(x, group, function(y) shapiro.test(y)))
Dále použijeme funkci lapply() a unlist() a melt() pro získání p-hodnot:
> sumstres<-lapply(stres, function(x) lapply(x, function(y) unlist(y)[2]))
> strespval<-melt(sumstres)
> strespval[which(as.numeric(as.character(strespval$value))<0.05),]
value L2 L1
9 0.000993783853791436 Cechy plodina5
16 0.0316488291795338 Morava plodina8
20 0.0136609485293158 Morava plodina10
Jak vidíme výnosy plodin 5, 8 a 10 nemají normální rozložení, proto provedeme testování pomocí Wilcoxnova testu, wilcox.test():
Vstupní hodnoty a skupiny můžeme definovat dvěma způsoby:
wilcox.test(hodnoty ve skupině 1, hodnoty ve skupině 2)
nebo funkčním zápisem:
wilcox.test(hodnoty~skupina)
Konkrétně pro otestování i-té plodiny (sloupce) by zápis vypadal takhle:
wilcox.test(uroda[group=="Cechy",i], uroda[group=="Morava",i])
nebo takhle
t.test(uroda[,i]~group)
> i <- 1
> vysledek <- wilcox.test(uroda[,i]~group)
> vysledek
Wilcoxon rank sum test with continuity correction
data: uroda[, i] by group
W = 12.5, p-value = 0.3217
alternative hypothesis: true location shift is not equal to 0
Jak získáme p-hodnotu? Funkce summary() ukáže všechny elementy výsledného objektu
> summary(vysledek)
Je mezi nimi i element s názvem p.value, hodnotu které získáme snadno:
> vysledek$p.value
[1] 0.3216665
Jedním ze základních výstupů je grafické zobrazení rozdílů mezi skupinami. Nejčastější je použití krabicového grafu (angl. boxplot). Všimněte si využití souboru plodiny pro extrakci názvu plodiny a funkce paste() pro konstrukci znakového řetězce kombinujícího tento název a p-hodnotu, který je použit v argumentu main=.
> boxplot(uroda[,i]~group, main=paste(plodiny[i,1], ", p-value=",round(vysledek$p.value,3)))
Jiným způsobem zobrazení je použití funkce stripchart(), která zobrazuje jednotlivá pozorování jako body ve skupinách
> stripchart(uroda[,i]~group, vertical=TRUE, method="jitter", jitter= 0.2, pch=19)
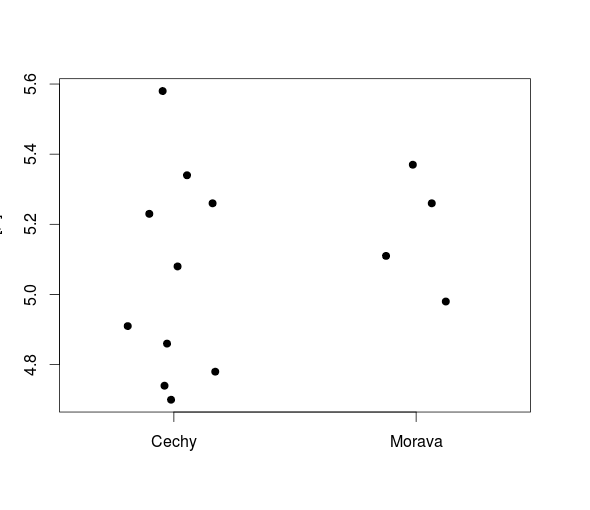
Chceme-li testovat hypotézu pro každou plodinu, aplikujeme funkci apply() na soubor uroda po řádcích. Z funkce wilcox.test() vybereme pouze p-hodnoty:
> vsechny.wtest <- apply(uroda, 2, FUN=function(x) wilcox.test(x~group)$p.value)
Dále chceme zjistit, rozdíly výnosů kterých plodin jsou statisticky významné, tedy které p-hodnoty jsou menší než naše zvolená hladina významnosti 0.05:
> vyznamne.wtest <- vsechny.wtest<0.05
> which(vyznamne.wtest)
plodina7
7
Při testování více hypotéz je potřeba korekce p-hodnot. Při testování 10 000 hypotéz na hladin významnosti 5 % totiž můžeme získat až 500 falešně pozitivních výsledků. Funkce pro korekci p-hodnot na mnohonásobné testování je p.adjust():
> p.adjust(vsechny.wtest, method = "bonferroni", n = length(vsechny.wtest))
plodina1 plodina2 plodina3 plodina4 plodina5 plodina6
1.0000000 1.0000000 1.0000000 1.0000000 0.7835429 1.0000000
plodina7 plodina8 plodina9 plodina10 plodina11 plodina12
0.4690480 1.0000000 1.0000000 0.7552448 1.0000000 1.0000000
plodina13 plodina14
1.0000000 1.0000000
Ve výsledku můžeme říct, že ani jedna z plodin nemá významně vyšší nebo nižší hektarové výnosy na Moravě nebo v Čechách.




